Dossier n°18, août 2006
Naissance d'une banque de données
par Séverine Altairac
![[ Naissance d'une banque de données ]](/prolune/images/prolune_cover018.jpg)
www.hadriendussoix.com
Interview du prof. Amos Bairoch
La banque de données de protéines Swiss-Prot fête son vingtième anniversaire. L'encyclopédie informatisée des protéines, aujourd'hui mondialement reconnue, a fait ses premiers pas en juillet 1986. Son histoire est indissociable de celle du prof. Amos Bairoch, son fondateur, qui est considéré comme l'un des pionniers de la bioinformatique : plein feux sur une aventure humaine peuplée de protéines ! [PDF] [english]
Amos Bairoch, à quoi sert une banque de données telle que Swiss-Prot et qu'est-ce que la bioinformatique?

Fig.1 Prof. Amos Bairoch
Swiss-Prot est destinée aux chercheurs en sciences de la vie. La banque de données présente, pour une protéine dans une espèce donnée, un résumé des informations disponibles, comme par exemple : où elle se trouve dans la cellule, comment elle est modifiée au cours du temps. L'information la plus importante demeure son rôle dans l'organisme. Si on ne sait pas à quoi sert une protéine, on peut s'en faire une idée en la comparant à une autre protéine qui lui ressemble. Swiss-Prot est donc un outil pour aider à caractériser des protéines nouvellement identifiées et dont on a déchiffré l'ordre des acides aminés qui les composent, c'est-à-dire leur séquence. Mais les informations condensées de Swiss-Prot ne remplacent pas les articles scientifiques au même titre qu'un article d'encyclopédie ne remplace pas la lecture des textes originaux.
La création et le maintien des banques de données font partie de la bioinformatique. On peut définir la bioinformatique comme la discipline de l'analyse de l'information biologique. Dès que l'on a commencé à accumuler des séquences d'ADN et de protéines, s'est posée la question de les stocker et de les comparer. Il fallait des ordinateurs. C'est ainsi qu'a débuté la bioinformatique, une quinzaine d'années après le séquençage de la première protéine - l'insuline en 1953. Les premiers outils développés furent des programmes d'analyse servant à comparer les protéines de différentes espèces et permettant ainsi l'étude de l'évolution.
Mais à cette époque la bioinformatique ne s'appelait pas encore ainsi ; le terme même est apparu il y a à peine 15 ans. On parlait alors d'analyse de séquences. Si on représente une protéine comme une suite de caractères, on peut alors l'analyser comme un texte en recherchant par exemple des lettres répétées ou des régions caractéristiques associées à des fonctions. D'autres zones, qui fonctionnent comme des étiquettes et permettent à la protéine de se diriger à un endroit précis dans la cellule, sont également repérables.
La bioinformatique s'est ensuite ouverte à d'autres types d'analyses comme celui de la forme que prend la protéine dans l'espace, c'est-à-dire sa structure tridimensionnelle.
Comment êtes-vous devenu bioinformaticien?

Fig.2 Premières passions du prof. Bairoch: astronomie et vie extraterrestre. Nébuleuse d'Orion
Je m'intéressais à la science-fiction, à l'astronomie et à la vie extraterrestre. J'avais lu beaucoup de choses sur comment détecter la vie, son apparition et son évolution, la fabrication des tout premiers acides aminés. Je me suis alors naturellement formé comme biochimiste, à l'université de Genève, car si on détecte un jour une vie extraterrestre, ce serait sans doute la discipline qui permettrait de l'étudier.
Ce qui me passionnait également, c'était les ordinateurs. Et celui qui m'a amené à la bioinformatique, bien qu'indirectement, est en fait Robin Offord. Robin est une grande pointure de la biochimie qui a travaillé aux côtés de Sanger lors des premiers séquençages protéiques. Je l'ai rencontré à l'occasion d'un job d'été dans un institut sur le diabète à Genève où j'avais proposé de programmer des logiciels permettant l'analyse des résultats expérimentaux obtenus par ce laboratoire. Puis, comme je commençais ma deuxième année de biochimie à l'université, je lui ai proposé de réaliser un programme qui mettrait bout à bout les différents fragments issus du séquençage d'une protéine, car lui à l'époque faisait l'assemblage à l'œil nu.
J'ai poursuivi ce projet lors de mon diplôme de master en biochimie dans le laboratoire-même de Robin. Il m'avait proposé de travailler sur l'analyse de protéines en utilisant un spectromètre de masse, une machine qui analyse les fragments d'une protéine après qu'elle a été découpée dans un tube à essai. Mais l'appareil est resté inutilisable pendant presque toute la durée de mon stage de diplôme ! Alors j'ai créé différents programmes pour analyser les séquences de protéines.
C'est finalement une passion conjointe pour les ordinateurs et pour les protéines qui m'a amené vers la bioinformatique. Ce qui est fascinant avec les protéines, c'est qu'elles agissent dans la cellule ou l'organisme, contrairement à l'ADN qui n'est que le support de l'information génétique.
D'où est née l'idée de créer une banque de données?
Au début de l'histoire des banques de données, les séquences de protéines étaient stockées dans un livre, l'Atlas of Protein Sequence and Structure de Margaret Dayhoff dont la première édition est parue en 1965 aux Etats-Unis. Puis ce répertoire est devenu, à la fin des années 70, la première banque informatisée NBRF/PIR -Protein Identification Resource. Les versions successives de cette banque de données étaient alors disponibles sur bande magnétique.
Alors que je développais PC/Gene, un ensemble de programmes d'analyse de séquences protéiques, je voulais utiliser la banque NBRF/PIR. Mais elle n'était pas faite pour un usage optimal sur ordinateur et j'ai entrepris de la convertir en un format plus adapté. Je me suis alors inspiré d'une banque d'ADN produite et maintenue par l'EMBL - European Molecular Biology Laboratory, en Allemagne. Nous avons alors augmenté le nombre initial de protéines contenues dans la banque NBRF/PIR avec des séquences saisies manuellement à partir d'articles scientifiques.
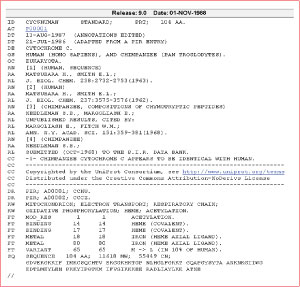
Fig.3 Format de Swiss-Prot à ses débuts
En reformatant NBRF/PIR, j'ai noté un certain nombre d'erreurs, d'oublis et de lacunes bibliographiques que j'ai signalés aux responsables de la banque de données. Mais jamais une seule réponse de leur part ne m'est parvenue. Je les ai même rencontrés lors d'une conférence aux USA en 1984 ; ils sont restés de marbre face à mes commentaires.
Alors, j'ai commencé à faire des corrections. Lorsque nous vendions les logiciels d'analyse PC/Gene, nous fournissions également la version corrigée de NBRF/PIR. Parce qu'elle était adaptée à une utilisation informatique, cette nouvelle version a rapidement séduit les utilisateurs. C'est pour répondre à ce besoin devenu évident, que j'ai décidé de créer une banque de données. Je l'ai nommée Swiss-Prot. La première version est sortie en juillet 1986. A ce moment-là, comme je voulais finir ma thèse et poursuivre le développement de PC/Gene, j'avais conclu un accord avec l'EMBL pour qu'ils prennent entièrement en charge la production et la mise à jour de Swiss-Prot dès la fin de l'année 86.
Mais l'avenir en a décidé autrement ! Les données sur les protéines ne cessaient de s'accumuler. Il fallait engager du personnel et le former. Bien que le groupe se soit agrandi avec l'arrivée de Rolf Apweiler, aujourd'hui directeur de l'European Bioinformatics Institute (EBI) à Cambridge, et de Brigitte Boeckmann à L'EMBL, puis avec l'arrivée de Serenella Ferro et de Jean-Pierre Patthey à Genève, l'effectif restait trop petit pour gérer la masse des données. Alors j'ai finalement décidé de continuer à faire évoluer Swiss-Prot !
Pourquoi avoir voulu un accès libre à Swiss-Prot sur Internet?
Aux débuts de Swiss-Prot, Internet n'existait pas. Les différentes versions étaient distribuées sur support magnétique moyennant les frais d'envoi. Avec l'avènement d'Internet, des systèmes d'interrogation à distance, puis du web, la banque est devenue totalement gratuite pour une meilleure accessibilité dans le monde entier.
Quelles sont les personnalités qui ont marqué votre "parcours bioinformatique"?
Robin Offord que j'ai rencontré pendant ma licence et qui m'a accueilli pour mon travail de master puis pour ma thèse. Il m'a donné carte blanche en me laissant acheter l'ordinateur dont j'avais besoin, m'a laissé commercialiser les logiciels d'analyse PC/Gene et exploiter les licences dans le but d'engager du personnel pour développer PC/Gene puis Swiss-Prot.
Mais surtout, Robin s'est battu contre vents et marées face à des informaticiens qui défendaient les ordinateurs et les terminaux alors gros comme des armoires. Les informaticiens de l'époque étaient en effet hostiles aux micro-ordinateurs, qui selon eux n'étaient bons que pour les jeux, du genre "space invaders"... Ils pensaient qu'ils ne seraient jamais utiles en science ... Dans les années 82-84, ce fut une terrible bagarre ! Bien que très puissants, les gros ordinateurs n'étaient pas adaptés à nos besoins. On ne voyait pas les résultats directement sur l'écran, pour ne citer qu'un exemple. Mais comme les micro-ordinateurs restaient hors de prix à l'époque, les commissions bloquaient des quatre fers toutes les commandes... Une des victoires de Robin a été d'obtenir l'achat du premier micro-ordinateur Apple II en Suisse ! Et c'était pour moi ...

Fig.4 Robin Offord et Jean-Michel Claverie
La deuxième personne à qui je dois beaucoup est Jean-Michel Claverie, que j'avais rencontré lors d'une réunion à l'EMBL. Nous sommes tout de suite devenus amis. Il était à l'Institut Pasteur de Paris et avait dans son équipe une jeune chercheuse, Lydie Bougueleret, qui aujourd'hui est devenue "mon bras droit" à Swiss-Prot. Le monde est petit !... Jean-Michel dirigeait l'Unité d'Informatique Scientifique de l'Institut Pasteur et nous avons décidé de faire ensemble une banque de données européenne. Malheureusement nous n'avons pas réussi à obtenir les fonds nécessaires. Par la suite, je lui ai demandé d'être mon directeur de thèse au niveau bioinformatique, tandis que Robin était mon directeur au niveau biochimie. Jean-Michel a accepté et j'ai pris régulièrement le train pour aller le voir à Paris.
A quel genre de difficultés vous êtes-vous heurté pour mener à bien le projet Swiss-prot?
Comme je l'ai déjà exposée précédemment : la réticence des informaticiens de l'époque aux micro-ordinateurs.
Nous nous sommes également heurtés à une autre difficulté de taille, celle de trouver des fonds privés. A l'époque, personne ne savait comment financer les banques de données. Pour l'anecdote, le projet que j'avais soumis avec Jean-Michel Claverie reposait sur le recrutement d'une seule personne. Les fonds européens nous avaient été refusés car une personne, c'était trop ! Quant au fonds national suisse, il soutenait des projets de recherche et non des infrastructures. Or une banque de données s'apparente davantage à une infrastructure qui fonctionne sur le long terme. Nous étions tout simplement face à une sorte d'inertie et il a fallu attendre la crise de 1996 pour faire bouger les choses.
Que s'est-il passé en 1996?
Le fonds national suisse avait accordé un financement pour 2-3 postes sur une durée de deux ans. Pour le renouveler, il nous a encouragés à faire une demande de fonds européen. Si le projet était accepté, il s'engageait alors à nous débloquer un nouveau financement.

Fig.5 Swiss-Prot menacée de disparition: annonce sur le serveur ExPASy en 1996
Nous avons donc soumis un dossier, conjointement avec l'EMBL qui nous soutenait aussi financièrement. Mais la demande a été refusée. La commission a reconnu la valeur scientifique de notre projet mais s'opposait à un financement supplémentaire à ceux de la Suisse et de l'EMBL. Mais en réalité, l'EMBL et le fonds national suisse attendaient l'aval de la commission européenne pour renouveler leur contrat avec nous. Le poisson se mordait la queue !
Avec Graham Cameron, à l'époque responsable de la banque de données à l'EMBL, nous avons contesté la décision prise à Bruxelles. Mais il était malheureusement impossible de faire marche arrière. On nous a suggéré de renouveler la demande une année plus tard. Mais nous n'avions pas les moyens de faire survivre Swiss-Prot et ses employés jusque là ! Nous avions juste deux mois devant nous pour trouver une solution ou plier boutique !
Comment a été sauvée Swiss-Prot?
En mai 96, nous avons fait un appel sur le serveur ExPASy, le site web qui héberge Swiss-Prot. Les emails ont commencé à affluer le jour-même. Nous avons reçu plus de 1000 emails et lettres de soutien. Tout cela a provoqué des réactions en chaîne : au niveau de la presse locale suisse, de la presse internationale scientifique (dans les revues Nature, Science) et au niveau de l'Union Européenne.

Fig.6 Amos Bairoch arborant les logos de l'Institut Suisse de Bioinformatique et de Swiss-Prot
En Suisse, le parlement fédéral a été soumis à une pression gigantesque. La ministre des sciences de l'époque, Ruth Dreyfus, a dû s'engager à mettre en place les mesures qui convenaient pour sauver Swiss-Prot le plus rapidement possible. En parallèle, le ministre de la santé du canton de Genève, Guy-Olivier Segond, a débloqué une somme permettant de payer les salaires jusqu'à la fin de l'année dans l'attente d'une solution fédérale. C'est fin 96 que le fonds national suisse nous a versé un financement d'urgence de deux ans en attendant de trouver une issue à plus long terme.
La solution à terme était la création de l'Institut Suisse de Bioinformatique. Il a fallu un peu moins de deux ans pour déterminer le cadre légal, faire les demandes de fonds, définir les statuts de l'institut et nommer un conseil de fondation. L'institut a bénéficié de l'article 16 de la constitution fédérale qui autorise la confédération à financer des recherches à but non lucratif et d'intérêt national. L'acte de naissance de l'institut a été signé en avril 98.
L'Institut Suisse de Bioinformatique est une confédération de groupes de recherche, il n'a pas de locaux. C'est un institut sans mur, "without wall" comme disent les Anglais. Les groupes se répartissent dans plusieurs villes, ce fut d'abord à Genève et Lausanne en 98, puis à Bâle et Zürich. L'institut vit grâce à un financement mixte. A la fois la confédération et les universités locales y contribuent.
Comment les personnes qui maintiennent la banque de données Swiss-Prot procèdent-ils?
Les personnes qui mettent à jour Swiss-Prot sont des annotateurs. Comme on annote un texte en y ajoutant des commentaires, les annotateurs introduisent des notes explicatives et informatives sur la structure et la fonction d'une protéine. Pour cela, ils se basent sur les articles scientifiques mais le point de départ de leur travail est la récupération des séquences de protéines.
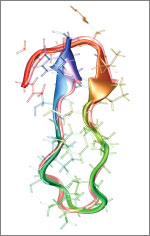
Fig.7 Outre les banques de données, la bioinformatique aide également à l'étude des structures protéiques (ici, la microcine J25)
Il y a 20 ans, les chercheurs étudiaient d'abord la fonction d'une protéine avant de la séquencer. Aujourd'hui on procède dans le sens inverse. Des groupes dans le monde entier déchiffrent des génomes entiers, c'est-à-dire l'ensemble de l'information génétique portée par l'ADN d'une espèce. L'ADN contient les gènes qui ne sont autres que les recettes de fabrication de protéines. Ces génomes portent l'information pour la fabrication de quelques centaines à quelques dizaines de milliers de protéines. Et toutes ces protéines virtuelles, dont l'existence n'a pas encore été prouvée expérimentalement, arrivent en masse dans les banques de données. Nous avons donc beaucoup de séquences protéiques à traiter mais avec beaucoup moins d'informations expérimentales sur la structure et la fonction des protéines qu'auparavant. Le travail d'analyse des annotateurs est donc aujourd'hui plus important.
Une conséquence directe de l'afflux massif de ces nouvelles séquences a été le problème de leur stockage. Ainsi a été développée, en 1996, une autre banque : TrEMBL gérée par l'EBI - European Bioinformatics Institute - à Hinxton. Les séquences ADN qui nous parvenaient étaient converties en séquences protéiques puis supprimées. Aujourd'hui elles sont stockées durablement dans la banque TrEMBL avant d'être analysées par les annotateurs de Swiss-Prot.
Au-delà de l'accumulation des séquences de protéines, les chercheurs accumulent aussi les résultats expérimentaux. Le travail de l'annotateur consiste aussi à suivre la littérature scientifique et à mettre perpétuellement à jour les données concernant une protéine. Une banque de données n'est jamais statique. En fonction des nouvelles découvertes, il faut en effet faire évoluer les informations.
Quel est l'atout de la banque Swiss-prot?
Sa qualité ! Chez Swiss-Prot, la qualité passe avant la quantité de protéines introduites dans la banque. Nous nous voulons précis sur les informations attribuées à chaque protéine. Pour s'affranchir de toutes les erreurs possibles, de la faute de frappe à l'imprécision scientifique, nous réalisons plusieurs niveaux de lectures et de vérification. L'idée est d'être aussi "précis qu'une horloge suisse" !...
Est-ce que certaines informations relatives aux protéines ont un intérêt médical?
C'est le cas des protéines humaines. La banque est un support à la recherche médicale dans le cadre de l'étude des maladies génétiques. De nombreux gènes peuvent subir un changement appelé mutation. Ceci se traduit parfois par un changement d'acide aminé dans la protéine correspondante, ce qui peut provoquer une maladie. Nous énumérons ces mutations et résumons les conséquences de ces modifications dans l'organisme.
C'est également le cas de certaines protéines bactériennes qui peuvent être des cibles pour le développement de nouveaux antibiotiques.
Quels ont été les grands changements qu'a connus Swiss-Prot?
La banque de données a connu deux grands changements. Le changement incontestablement le plus grand est l'accès au texte des publications scientifiques directement sur Internet. Il y a encore 5-6 ans, nous devions aller photocopier les articles en bibliothèque. Ça a changé la vie des annotateurs !
Le deuxième changement concerne la spécialisation de l'annotateur. Aux débuts de Swiss-Prot, l'annotateur était un généraliste. Un jour il travaillait sur une protéine bactérienne, le lendemain sur une protéine humaine. Parce que la masse d'informations et leur complexité a augmenté, nous avons dû spécialiser le travail d'annotation. Par exemple, pour travailler sur des protéines virales, nous avons engagé un virologiste.
Pour quelles espèces dispose-t-on aujourd'hui de toutes les protéines dans Swiss-prot?
Il y en a plusieurs. Les virus par exemple car ils possèdent peu de protéines. Le virus du SIDA en contient dix. On a également toutes les protéines de Escherichia coli - plus de 4000, la bactérie la plus connue. A la fin de l'année, on aura répertorié toutes les protéines de la levure de boulanger, Saccharomyces cerevisiae, soient 6000 protéines.
Et pour quand, toutes les protéines humaines?
Le séquençage du génome humain est fini. On estime entre 20'000 et 25'000 le nombre de gènes et à 500'000 celui des protéines chez l'homme. Dans cette estimation, on exclut les anticorps, des protéines de la défense immunitaire, qui sont fabriqués spécifiquement pour répondre à une situation particulière et dont le nombre est sans aucun doute faramineux, certainement de plusieurs millions.
A quand toutes les protéines humaines dans Swiss-Prot ? On peut espérer en avoir la totalité annotée, au moins de façon sommaire, d'ici deux ans...
Quelle est la dimension qu'a prise Swiss-prot?
En 2002, les instituts nationaux de la santé des USA -les NIH- ont lancé un appel d'offre pour développer une banque de données unifiée de protéines. L'Institut Suisse de Bioinformatique, l'European Bioinformatics Institute et la banque de données américaine PIR ont décidé d'y répondre dans un projet commun. Ensemble nous avons créé un consortium qui s'appelle UniProt dont le but est de continuer le développement des banques de données en perpétuant la collaboration entre ces trois instituts. Le projet a été accepté fin 2002, pour une période de 3 ans et a été reconduit une deuxième fois.
Quel avenir pour Swiss-Prot?
Il est difficile de prévoir vers quoi tend Swiss-Prot. Il s'est toujours passé des évènements imprévisibles. En tout cas, il y aura toujours de plus en plus d'informations, de plus en plus de séquences et de plus en plus de travail à accomplir. En vingt ans, le nombre de protéines entrées dans la banque de données est passé de 4'000 à plus de 230'000 et l'effectif de Swiss-Prot de une à plus de 70 personnes. Parmi les annotateurs, certains partagent leur savoir et leurs compétences depuis plus de quinze ans. Malgré la qualité de leur travail à tous, ils ne seront certainement pas assez nombreux pour assurer la maintenance dans le futur.
Peut-être d'autres pays se joindront-ils à nous plus tard. Il y a déjà un groupe au Brésil qui nous aide et un groupe japonais va bientôt rejoindre UniProt. C'est une manière d'accroître les possibilités de financement et également de diversifier les compétences des annotateurs, une combinaison qui permettra sans doute à Swiss-Prot d'assurer son avenir...


